Analýza fosfoproteínov a signálnych dráh kvantitatívno- proteomickými metódami
|
|
|
- Shawn Long
- 5 years ago
- Views:
Transcription
1 Analýza fosfoproteínov a signálnych dráh kvantitatívno- proteomickými metódami Ananlysis of Phosphoproteins and Signalling Pathways by Quantitative Proteomics Pjechová M. 1, Hernychová L. 1, Tomašec P. 1,2, Wilkinson G. W. 1,2, Vojtěšek B. 1 1 Regionální centrum aplikované molekulární onkologie, Masarykův onkologický ústav, Brno 2 Department of Medical Microbio logy, Institute of Infection and Immunity, School of Medicine, Cardiff University, United Kingdom Súhrn Fosforylácia proteínov má kľúčovú úlohu v regulácii bunkových signálnych dráh. Je zahrnutá vo väčšine bunkových dejov, v ktorých súhra medzi kinázami a fosfatázami prísne kontroluje bunkové deje ako napr. proliferáciu, diferenciáciu a apoptózu. Chybné alebo pozmenené signálne dráhy sa mnohokrát podieľajú na vzniku rôznych chorôb, čo iba zdôrazňuje dôležitosť štúdia fosfoproteómu. Abundancia fosfoproteínov je v proteóme často veľmi nízka a na ich analýzu sú potrebné vysoko citlivé a špecifické prístupy. Metódami kvantitatívnej proteomiky je možné analyzovať zmeny v abundancii jednotlivých proteínov a ich posttranslačných modifikáciách a následne i v signálnych dráhach buniek. V tomto článku sa venujeme kvantitatívno- proteomickým metódam, ktoré je možné použiť pri štúdiu fosfoproteínov a ich zapojení do signálnych dráh. Kľúčové slová proteomika fosfoproteíny signálne dráhy Summary Protein phosphorylation is a key regulator in cellular signaling pathways. It is involved in most cellular events in which interplay between phosphatases and kinases strictly controls bio logical processes, such as differentiation, proliferation and apoptosis. Altered or defective signaling pathways often result in various diseases, emphasizing the importance of studying the phosphoproteome. The abundance of phosphoproteins in the proteome is often very low, which requires specific and highly sensitive approaches. By using quantitative proteomics methods, we are able to analyze changes in abundance of proteins and their posttranslational modifications and then changes in signaling pathways. In this review, we describe quantitative proteomics methods, which could be used for study of phosphoproteins and their connection in signaling pathways. Key words proteomics phosphoproteins signaling pathways Práca bola podporená Európskym fondom pre regionálny rozvoj a štátnym rozpočtom Českej republiky (OP VaVpI RECAMO, CZ.1.05/2.1.00/ ) a MZ ČR RVO (MOÚ, ). This work was supported by the European Regional Development Fund and the State Budget of the Czech Republic (RECAMO, CZ.1.05/2.1.00/ ) and by MH CZ DRO (MMCI, ). Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy. The authors declare they have no potential conflicts of interest concerning drugs, products, or services used in the study. Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do bi omedicínských časopisů. The Editorial Board declares that the manuscript met the ICMJE uniform requirements for biomedical papers. Mgr. Mariana Pjechová Regionální centrum aplikované molekulární onkologie Masarykův onkologický ústav Žlutý kopec Brno mariana.pjechova@mou.cz Obdržené/Submitted: Přijaté/Accepted: S116
2 Úvod Fosforylácia proteínov je jedným z najrozšírenejších regulačných mechanizmov v prírode. Je kľúčovým regulátorom vnútrobunkových bio logických procesov a momentálne najštudovanejšou posttranslačnou modifikáciou proteínov. Fosforyláciou je obvykle iniciovaná signálna transdukcia a šírené signály v signálnych dráhach buniek. Je to dočasná a reverzibilná posttranslačná modifikácia, ktorá vedie k zmenám konformácie, aktivity a interakcií proteínov vo veľmi krátkom časovom úseku. Proteínovú fosforyláciu riadia dve skupiny proteínov: kinázy (fosforylujú proteíny) a fosfatázy (defosforylujú proteíny). Často sú fosforylované viaceré reziduá proteínu, a to väčšinou rôznymi kinázami. Spomedzi aminokyselín sa u eukaryotov môže fosforylovať serín, treonín, tyrozín a histidín, ktorý je však veľmi labilný a zriedka identifikovaný vo fosfoproteomických štúdiách. Prístupy kvantitatívnej proteomiky nám dovoľujú analyzovať zmeny v expresii proteínov (a fosfoproteínov) za rôznych podmienok a taktiež aj určiť zmenu fosforylácie jednotlivých proteínov, ktoré tvoria základnú funkčnú jednotku signálnych dráh. V porovnaní s ostatnými proteínmi sú fosfoproteíny v bunkách prítomné vo veľmi nízkych množstvách, čo komplikuje ich detekciu hmotnostným spektrometrom a následné analýzy. Z tohto dôvodu sú potrebné techniky, ktoré umožňujú obohatenie vzoriek o fosfopeptidy (po proteolytickom štiepení proteínov na peptidy) pred hmotnostne spektrometrickou analýzou. Strong cation exchange chromatography (SCX) [1], hydrophilic interaction liquid chromatography (HILIC) [2], obohatenie fosfopeptidov oxidom titaničitým (TiO2) [3] či immobilized metal ion affinity chromatography (IMAC) [1] patria medzi techniky pomocou ktorých sme schopní úspešne izolovať fosfopeptidy pred MS analýzou. Následná kvapalinová chromatografia v spojení s tandemovou MS analýzou (LC- MS/ MS) je vhodný prístup, ktorý dovoľuje identifikáciu a kvantifikáciu tisícok fosforylačných miest počas jednej analýzy. Tieto, ale aj ďalšie metódy a postupy používané vo fosfoproteomických experimentoch budeme bližšie rozoberať v nasledujúcej časti článku. Metódy kvantifikácie fosfoproteínov Na kvantifikáciu zmien fosfoproteínov je možné použiť proteomické prístupy ako napr. stable isotope labeling with amino acids in cell culture (SILAC) [4], ktorý sa dá použiť iba na živé bunkové kultúry. Jedná sa o metabolické značenie aminoyselín, pri ktorom v priebehu kultivácie dochádza k inkorporácii ťažkých aminokyselín (značené stabilné izotopy lyzínu a arginínu) do novosyntetizovaných proteínov. Pri tejto metóde je možné použiť ťažké izotopy lyzínu a arginínu (R10K8), stredne ťažké izotopy lyzínu a arginínu (R6K4) a neznačené, ľahké médium (R0K0). Medzi izotopické značenia fosfopeptidov patria napr. ICAT, dimetylové a 18 O značenie. ICAT (isotope- coded affinity tags), teda thiol- reaktívne molekuly obsahujúce ťažké alebo ľahké izotopy, predstavuje jednu z najstarších techník [5]. ICAT značka pozostáva z reaktívnej skupiny, ktorá je orientovaná na cysteín, polymerového linkera s ôsmimi deutériami a bio tínovej skupiny, ktorá dovoľuje purifikáciu značených peptidov. V ICAT experimente sú dva proteínové pooly denaturované a redukované, cysteínové reziduá proteínov sú následne značené ľahkým alebo ťažkým reagentom. Značené pooly sú potom zmiešané, prečistené od nadbytečného reagentu a štiepené vhodnou proteázou. Peptidy obsahujúce cysteín, nesúce ľahkú alebo ťažkú značku sú následne zachytené pomocou avidínovej kolóny, cez bio tínovú skupinu na značke. Peptidy sú potom vyeluované z kolóny a analyzované na MS [6]. Pri dimetylovom značení sú proteíny najprv štiepené vhodnou proteázou, ako napr. trypsín, následne sú peptidy z rôznych vzoriek označené dimetylovými značkami a analyzované na LC/ MS, pričom rozdiel abundancie dimetylových značiek je použitý na porovnanie rozdielov v abundancii peptidov vo vzorkách. Toto značenie je založené na reakcii primárnych peptidových amínov (N- konce peptidov a epsilonové amino skupiny lyzínových reziduí) s formaldehydom a vytvorení Schiff bázy, ktorá je rapídne redukovaná pridaním cyanoborohydridu. Dimetylové značenie je možné použiť ako triplex, kde je pri značení ľahkou značkou použitý obyčajný formaldehyd a cyanoborohydrid, čo spôsobí zvýšenie hmotnosti o 28 Da na primárny amín v peptide. Použitie denaturovaného formaldehydu a obyčajného cyanoborohydridu spôsobí zvýšenie hmotnosti o 32 Da, čo je stredná značka [7]. Inkorporácia ťažkej značky je dosiahnutá kombináciou denaturovaného a 13 C- značeného formaldehydu s cyanoborodeuteridom, čo vedie k zvýšeniu hmotnosti o 36 Da [8]. Značenie pomocou 18 O je relatívne jednoduchá, enzýmovo- sprostredkovaná technika značenia peptidov pomocou H 2 18 O. Tento prístup je spojený s proteázami 2. triedy, napr. trypsínom, ktoré katalyzujú výmenu dvoch 16 O atómov za dva 18 O atómy na C-terminálnej karboxylovej skupine proteolitického peptidu, čo vedie k posunu hmotnosti o 4 Da medzi 1- krát nabitými rôzne značenými peptidmi v MS1 móde [6]. TMT (tandem mass tag) [9] a itraq (the isobaric tag for relative and absolute quantitation) [10] sú izobarické značenia fungujúce na veľmi podobnom princípe. V porovnaní s tradičnými kvantifikačnými metódami sú schopné použitím tandemovej MS analýzy simultánne identifikovať a kvantifikovať 6 8 vzoriek. TMT molekula pozostáva z reaktívnej skupiny, ktorá značí primárne amínové skupiny; reportérovej skupiny, ktorá udáva v MS/ MS móde abundanciu peptidu po zmiešaní peptidov; odštiepiteľného linkeru, ktorý uvoľnuje TMT reporterový fragment v MS/ MS analýze a hmotnostne normalizačnej skupiny, ktorá vyrovnáva hmotnostné rozdiely medzi jednotlivými reportérovými fragmentami na udržanie konštantnej celkovej hmotnosti značiek v sete [11]. Keďže sa jedná o izobarické značky, kvantifikácia prevedená v MS/ MS móde porovnaním plôch píkov označených iónov vychádza z fragmentácie značky, teda rovnaké spektrum je použité na kvantifikáciu a identifikáciu fosfopeptidu [12]. S117
3 Pri label free kvantifikácii sú proteínové zmesi analyzované priamo a vzorky sú navzájom porovnané až po samostatných analýzach. Pri tejto metóde nie sú vzorky zmiešané, teda je možné dosiahnuť vyššieho proteómového pokrytia a neexistuje žiaden limit v počte vzoriek, ktoré môžu byť porovnávané [13]. Nevýhodou tohto prístupu je neprítomnosť formálneho vnútorného štandardu, čo môže viesť k významným chybám v individuálnych datasetoch. Tento nedostatok je však minimalizovaný analýzou niekoľkých bio logických replikátov [6]. Proteolytické štiepenie Pred hmotnostnou analýzou je potrebné vzorky upraviť tak, aby boli kompatibilné s meraním na hmotnostnom spektrometri. Prvým krokom je štiepenie proteínov na peptidy použitím vhodnej proteázy. Na to, aby mohla proteáza proteíny efektívne štiepiť, je potrebná ich denaturácia, redukcia a alkylácia. Na denaturáciu sa používajú rôzne reagencie, napr. β-merkaptoetanol, SDS. Medzi najviac používané redukčné činidlo patrí dithiotreitol (DTT) alebo Tris (2- carboxyethyl) phosphine (TCEP), ktoré redukujú disulfidové mostíky medzi cysteínovými skupinami. Nasleduje alkylácia thiolových skupín pomocou jódacetamidu (IAA), ktorý zabraňuje ich reoxidácii. Proteíny sú potom štepené proteázami na peptidy, ktoré je možné analyzovať na hmotnostnom spektrometri. Používajú sa proteázy s vysokou špecificitou štiepenia, aminokyselinové reťazce štiepia iba za určitými aminokyselinami. Jednou z najbežnejšie používaných proteáz je trypsín, ktorý štiepi reťazec aminokyselín na karboxylovej strane lyzínu a arginínu, ak za nimi nenasleduje prolín. Existuje viacero postupov, ako proteíny pripraviť na proteolytické štiepenie. Ich výber závisí na type a množstve vzorky, keďže pri niektorých postupoch sú starty vzoriek väčšie než pri iných. Pred vlastným proteolytickým štiepením je potrebné odstrániť denaturačný roztok, ktorý nie je kompatibilný s proteolytickými enzýmami, a preto sa zamieňa za roztok močoviny. Pri filter aided sample preparation (FASP) protokole sa tento krok deje na cut off fil- tri (10 kda, alebo 30 kda), kde prebieha aj následná redukcia, alkylácia a vlastné proteolytické štiepenie. Medzi nevýhody tohto postupu patrí čiastočná strata vstupného materiálu na filtri [14]. Štiepenie v roztoku je postup, pri ktorom sa do skúmavky so vzorkou proteínov postupne pridávajú jednotlivé roztoky zaisťujúce denaturáciu, redukciu a alkyláciu (napr. močovina, DTT, IAA). Prebytok alkylačného činidla je následne odstránený pridaním redukčného činidla, aby sa v hmotnostnom spektre neobjavovali satelitné peptidové píky nesúce vzniknuté modifikácie zvyšujúce ich m/ z. Nakoniec sa pridáva proteáza a roztok NH 4 HCO 3, ktorý je s ňou kompatibilný a poskytuje vhodné podmienky na štiepenie. V prípade tohto postupu vstupný materiál síce zostáva zachovaný, ale všetky pridané reagencie zostávajú v roztoku spolu so vzorkou a pred ďalším krokom je nutné získané peptidy purifikovať [15]. Pri štiepení proteínov v géli sa bunkový lyzát najprv rozseparuje na polyakrylamidovom géli. Zväčša sa používajú denaturačné 1D alebo 2D elektroforézy, ktoré separujú denaturované proteíny podľa molekulovej hmotnosti (proteíny s najvyššou hmotnosťou putujú v géli najpomalšie), čím zároveň umožňujú presné stanovenie molekulovej hmotnosti. Pri 1D elektroforéze sa proteíny separujú iba podľa molekulovej hmotnosti. Pri 2D elektroforéze sa proteíny rozdeľujú najprv podľa izoelektrického bodu, čo je ph, pri ktorom majú molekuly nulový náboj, a nepohybujú sa v elektrickom poli [16]. Nasleduje ich delenie v druhom smere podľa molekulovej hmotnosti. Najväčšou výhodou týchto postupov je prečistenie proteínov od nízkomolekulových nečistôt, ako napr. detergentov a zložiek pufrov, ktoré často nie sú kompatibilné s následnou MS analýzou. Na vizualizáciu proteínov sa používajú napr. trifenylmetánové farbivá Coomassie brilliant blue (R250, G250), ktoré sa viažu na proteíny prostredníctvom van der Waalesových a elektrostatických interakcií [17]. Najbežnejšie používanou je Coomassie brilliant blue G250, ktorá je schopná vizualizovať už 0,3 ng proteínu v géli. Komerčne sú dostupné aj fluorescenčné značky, napr. Sypro Ruby, Flamingo, či Deep Purple, ktoré majú senzitivitu okolo 1 ng detekovaného proteínu. Veľmi citlivým prístupom je farbenie striebrom, pomocou ktorého je možné vizualizovať 0,3 ng proteínu. Po farbení sú jednotlivé bandy vyrezané z gélu, odfarbené a proteíny štiepené proteázou. Nevýhoda tejto metódy spočíva v strate proteínov nerozpustných v SDS pufri a proteínov s nízkou molekulovou hmotnosťou [18]. Separácia peptidov pomocou kvapalinovej chromatografie Na rozdelenie komplexných vzoriek peptidov získaných FASP protokolom alebo štiepením v roztoku, ako aj na odstránenie zložiek, ktoré môžu interferovať s ďalšími postupmi, je možné použiť kvapalinovú chromatografiu. High-performance liquid chromatography (HPLC) môže byť použitá na separáciu peptidov na základe hydrofobicity, náboja alebo veľkosti. Kombináciou vhodných analytických kolón a gradientov je možné vybrať frakcie obsahujúce peptidy a fosfopeptidy a ďalej ich analyzovať. Vybrané typy kolón a ich vlastnosti budú popísané v ďalšej časti: Na negatívne nabitých funkčných skupinách kolóny strong cation exchange chromatography (SCX) sa pri ph 2,7 zachytávajú najmä 1- krát fosforylované +1 kladne nabité fosfopeptidy. V eluovanej frakcii sa nachádzajú multifosforylované peptidy, preto je nutné eluát zachytiť [1]. Hydrophilic interaction liquid chromatography (HILIC) je vysoko rozlišovacia separačná technika, pri ktorej sa využívajú vodíkové interakcie medzi peptidom a neutrálnou, hydrofilnou stacionárnou fázou. Retencia vzrastá so zvyšujúcou sa polaritou peptidu. Vzorky sú nanášané vo fáze s vysokým podielom organických zlúčenín a eluo vané zvyšujúcou sa polaritou mobilnej fázy [2]. Electrostatic repulsion- hydrophilic interaction chromatography (ERLIC) je separácia založená na anion exchange chromatography v kombinácii s hydrofilnými interakciami. Fosfátové skupiny sú čiastočne ionizované a elektrostaticky priťahované k stacionárnej S118
4 Hmotnostne spektrometrická analýza Peptidy (fosfopeptidy) je možné identifikovať pomocou MALDI- MS (matrix- aassis ted laser desorption/ ionization mass spectrometry, tj. hmotnostného spektrometru s ionizáciou pomocou laseru za účasti matrice) alebo prostredníctvom LC- MS/ MS (liquid chromatography mass spectrometry) s elektrosprejovou ionizáciou. Pri hlbších fosforylačných štúdiách je dôležité určiť špecifickú aminokyselinu, ktorá bola fosforylovaná (miesto fosforylácie) v danom bio logickom experimente. Preto sa využíva tandemová MS analýza s collision-induced dissociation (CID) a electron- transfer dissociation (ETD) fragmentáciou. Pri CID fragmentácii dochádza k strate fosfátovej skupiny H 3 PO 4 pri fosfoseríne a fosfotreoníne a HPO 3 pri fosfotyrozíne. Nedostatkami tejto metódy sú nízka ionizačná efektivita a skorá strata labilnej fosfátovej skupiny, čo má za následok nízky počet peptidových fragmentov na identifikáciu. ETD štiepi peptidovú kostru bez odštiepenia labilných posttranslačných mofáze. Ako prvé sú eluované nefosforylované, monofosforylované a difosforylované peptidy, keďže multifosforylované peptidy obsahujú viacero negatívnych nábojov na fosfátových skupinách a eluujú sa neskôr [19]. Techniky obohacovania vzoriek o fosfopeptidy Obohatenie fosfopeptidov je kľúčový krok v príprave vzoriek na identifikovanie fosforylovaných aminokyselín. Koncentrácia fosfoproteínov je v bunke v porovnaní s ostatnými proteínmi veľmi nízka a sú takmer nemerateľné dostupnými technikami hmotnostnej spektro metrie. Fosfopeptidy zle ionizujú, čo je dôsledkom prítomnosti fosfátovej skupiny, ktorá je záporne nabitá. Tým ovplyvňuje efektivitu ionizácie a detekcie pri kyslých podmienkach, ktoré sa používajú pri analýze v pozitívnom móde [20]. Fosfopeptidy bývajú v spektrách prekryté fragmentmi nefosforylovaných/ abundantnejších peptidových iónov, ktoré majú oveľa vyššiu intenzitu signálu. Na získanie/ obohatenie fosfopeptidov zo vzoriek bolo vyvinutých viacero metód, ktoré využívajú väzbu fosfátovej skupiny na matricu. Medzi najviac používané obohacovacie metódy patria obohatenie fosfopeptidov oxidom titaničitým (TiO 2 ), immobilized metal ion affinity chromatography (IMAC) a sequentional elution from IMAC (SIMAC). V prípade TiO 2 (voľné častice s priemerom 10 μm) dochádza k väzbe fosfopeptidu cez fosfátové OH skupiny za kyslých podmienok. Uvoľnenie tejto väzby nastáva za zásaditých podmienok, pričom uvoľnené sú najmä monofosforylované peptidy (multifosforylované peptidy sa na TiO 2 viažu s príliš vysokou afinitou a ich elúcia je veľmi obtiažna). Na TiO 2 sa môžu viazať aj kyslé peptidy, čomu sa dá predísť znížením ph kyselinou trifluóroctovou alebo pridaním kyseliny dihydrobenzoovej, či mliečnej, ktoré sa na TiO 2 viažu s vyššou afinitou než kyslé peptidy, ale s nižšou než fosfopeptidy [3]. Pri metóde IMAC sú ióny kovov (Fe 3+, Al 3+, Ga 3+, Co 2+ ) chelatované na častice pokryté nitrilotrioctovou alebo iminodioctovou kyselinou, čím vytvárajú sta- cionárnu fázu, na ktorú sa môžu naviazať negatívne nabité fosfopeptidy v mobilnej fáze. Táto metóda je vhodná hlavne pre obohatenie multifosforylovaných peptidov. Vážnym problémom tohto postupu je nešpecifická väzba nefosforylovaných peptidov obsahujúcich viacnásobne kyslé reziduá aminokyselín, čo znižuje selektivitu. Jednou z možností ako zvýšiť selektivitu tejto metódy je rovnako ako pri TiO 2 zníženie ph [1]. SIMAC metóda kombinuje výhody TiO 2 a IMAC prístupu. Keďže pri IMAC sú preferenčne naviazané multifosforylované peptidy a pri TiO 2 monofosforylované, kombináciou týchto dvoch metód je možné dosiahnuť jednoduchého obohatenia mono- i multifosforylovaných peptidov. IMAC je použitý ako prvý krok, kde po väzbe nasleduje elúcia monofosforylovaných peptidov za kyslých podmienok (ph 1,0) a potom elúcia multifosforylovaných peptidov za bázických podmienok (ph 11,3). Ďalším krokom je obohatenie monofosforylovaných peptidov TiO 2 časticami [1]. difikácií, ako napr. fosforylácie. ETD má vyššiu pravdepodobnosť úspešnej fosfopeptidovej identifikácie pre prekurzory s nízkym m/ z a nábojovým stavom > 2, kým CID je úspešnejšia pre dvojnásobne nabité peptidy a tie s vysokým m/ z [21]. Niektoré štúdie porovnávajúce ETD a CID navrhujú využitie oboch metód fragmentácie na dosiahnutie komplexnej analýzy fosforylácie [21 23]. Analyzovanie dát Kritickou súčasťou všetkých proteomických štúdií je analýza dát, pri ktorej sa využívajú bio informatické a štatistické prístupy. Získané MS/ MS dáta sú porovnané s databázou Uniprot proteome [24] s použitím softwarov Mascot (Matrix Science Inc.) alebo Sequest [25]. Na spracovanie dát, prehľadanie databáz a kvantitatívne vyhodnotenie rozdielov medzi fosfoproteínmi v jednotlivých vzorkách je možné použiť program Proteome Discoverer 1.4 (Thermo Fisher Scientific) s algoritmom PhosphoRS 3.1 [26] umožňujúcim predikciu lokalizácie fosforylovaných miest. K dispozícii sú aj ďalšie podobné programy MaxQuant [27] a SkyLine [28]. Pri identifikovaných fosfoproteínoch je potrebné brať do úvahy zmenu fosforylácie oproti kontrolnej vzorke a celkovej expresii proteínu. Proteíny so signifikantnými zmenami fosforylácie môžu byť ďalej použité na štúdium signálnych dráh pomocou špecializovaných programov ako napr. Ingenuity Pathway Analysis (IPA) [29], David [30] alebo PathVisio [31]. Tieto programy sú schopné graficky znázorniť signálne dráhy na základe informácií získaných z databáz a vedeckých publikácií. Umožňujú tiež porovnanie jednotlivých dráh, a teda definovanie hlavných rozdielov medzi signálnymi dráhami v rôznych bunkách. Záver Na štúdium zmien v signálnych dráhach buniek je možné využiť proteomické prístupy. Kvantitatívna proteomika umožňuje určiť zmeny v signálnych dráhach zahŕňajúce zmeny v expresii proteínov, ako aj zmeny v posttranslačných modifikáciách (fosforyláciách), ktoré S119
5 Proteolitické štiepenie a) FASP filter aided sample preparation b) štiepenie v roztoku c) štiepenie v géli Kapalinová chromatografia a) SCX strong cation exchange b) HILIC hydrophilic interaction liquid chromatography c) ERLIC electrostatic repulsion hydrophilic interaction chromatography Obohatenie fosfopeptidov a) TiO 2 b) IMAC immobilized metal ion affinity chromatography c) SIMAC sequential elution from IMAC Hmotnostne spektrometrická analýza a) MALDI/MS b) LC-ESI/MS + tandemové MS/MS analýzy (CID, ETD) Analýza dát určenie signálnych dráh a) ingenuity pathway analysis b) path visio c) david Tab. 1. Schéma postupu spracovania vzorky proteomickými metódami. sú základným mechanizmom pri zapínaní/ vypínaní signálnych dráh. Vzhľadom na nízku abundanciu fosfoproteínov v bunkách a ich problematické zachytenie hmotnostným spektrometrom je správna voľba metód kľúčovým krokom (tab. 1.). Nedostatky pri momentálne používaných postupoch poskytujú široký priestor na vylepšenie metód. Zamedzenie straty materiálu pri príprave vzoriek, či väzbe kyslých peptidov na častice pri obohacovacích technikách sú problémy, ktoré je nutné riešiť. Nemenej dôležitými sú i obmedzenia v MS analýze a bio informatických aplikáciách. Rovnako aj zdokonaľovanie starých a vývoj nových metód a programov na analýzu môže rapídne prispieť k vylepšeniu detekcie a identifikácie proteínov. Literatúra 1. Thingholm TE, Jensen ON, Larsen MR. Analytical strategies for phosphoproteomics. Proteomics 2009; 9(6): doi: / pmic Alpert AJ. Hydrophilic- interaction chromatography for the separation of peptides, nucleic acids and other polar compounds. J Chromatogr 1990; 499: Larsen MR, Thingholm TE, Jensen ON et al. Highly selective enrichment of phosphorylated peptides from peptide mixtures using titanium dioxide microcolumns. Mol Cell Proteomics 2005; 4(7): Oda Y, Huang K, Cross FR et al. Accurate quantitation of protein expression and site- specific phosphorylation. Proc Natl Acad Sci USA 1999; 96(12): Gygi SP, Rist B, Gerber SA et al. Quantitative analysis of complex protein mixtures using isotope- coded affinity tags. Nature Biotechnol 1999; 17: Sap KA, Demmers JA (eds). Labeling Methods in Mass Spectrometry Based Quantitative Proteomics [monograph on the Internet]. Integrative Proteomics; 2012 [cited 2014 January 3]. Available from: / cdn.intechopen. com/ pdfs- wm/ pdf. 7. Hsu JL, Huang SY, Chow NH et al. Stable- isotope dimethyl labeling for quantitative proteomics. Anal Chem 2003; 75(24): Boersema PJ, Aye TT, van Veen TA et al. Triplex protein quantification based on stable isotope labeling by peptide dimethylation applied to cell and tissue lysates. Proteomics 2008; 8(22): doi: / pmic Thompson A, Schafer J, Kuhn K et al. Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/ MS. Anal Chem 2003; 75(8): Ross PL, Huang YN, Marchese JN et al. Multiplexed protein quantitation in saccharomyces cerevisiae using amine- reactive isobaric tagging reagents. Mol Cell Proteomics 2004; 3(12): Licker V, Patel V, Ward M (eds). Characterisation of human cerebrospinal fluid (CSF) after tandem mass tag (TMT0) labelling [monograph on the Internet]. Universtité de Genéve; 2008 [cited 2014 January 3]. Available from: / reports/ rap_virginielicker.pdf. 12. Nita- Lazar A, Saito- Benz H, White FM. Quantitative phosphoproteomics by mass spectrometry: past, present, and future. Proteomics 2008; 8(21): doi: / pmic Bantscheff M, Schirle M, Sweetman G et al. Quantitative mass spectrometry in proteomics: a critical review. Anal Bioanal Chem 2007; 389(4): Wiśniewski JR, Zougman A, Nagaraj N et al. Universal sample preparation method for proteome analysis. Nat Methods 2009; 6(5): doi: / nmeth Btk.fi [homepage on the Internet]. Turku: Centre for Biotechnology, Finland [cited 2014 January 5]. Avail able from: / fileadmin/ Page_files/ proteomics/ PDF- files/ Protocols/ In_solution_trypsin_digestion. pdf. 16. Vgn.uvm.edu [homepage on the Internet]. Vermont Genetics Network. University of Vermont [cited 2014 January] Available from: / vgn.uvm.edu/ outreach/ documents/ Garfin_IEF_WebArticle9 07.pdf. 17. Nationaldia gnostics.com [homepage on the Internet]. Atlanta: National Dia gnostics; Georgia; c2011 [cited 2014 January 5]. Available from: / electrophoresis/ article/ staining- protein-gels- coomassie- blue. 8. Shevchenko A, Tomas H, Havlis J et al. In- gel digestion for mass spectrometric characterization of proteins and proteomes. Nat Protoc 2006; 1(6): Alpert AJ. Electrostatic repulsion hydrophilic interaction chromatography for isocratic separation of charged solutes and selective isolation of phosphopeptides. Anal Chem 2008; 80(1): Steen H, Jebanathirajah JA, Rush J et al. Phosphorylation analysis by mass spectrometry. Mol Cell Proteomics 2006; 5(1): Swaney DL, McAlister GC, Coon JJ. Decision tree-driven tandem mass spectrometry for shotgun proteomics. Nat Methods 2008; 5(11): doi: / /nmeth Molina H, Horn DM, Tang N et al. Global proteomic profiling of phosphopeptides using electron transfer dissociation tandem mass spectrometry. Proc Natl Acad Sci USA 2007; 104(7): Sweet SM, Bailey CM, Cunningham DL et al. Large scale localization of protein phosphorylation by use of electron capture dissociation mass spectrometry. Mol Cell Proteomics 2009; 8(5): doi: / mcp. M MCP Uniprot.org [homepage on the Internet] Uniprot, c [cited 2014 January 5]. Available from: / Eng JK, McCormack AL, Yates JR. Sequest algorithm publication: an approach to correlate tandem mass spectral data of peptides with amino acid sequences in a protein database. J Am Soc Mass Spectrom 1994; 5(11): doi: / (94) Taus T, Köcher T, Pichler P et al. Universal and confident phosphorylation site localization using phosphors. J Proteome Res 2011; 10(12): doi: / pr200611n. 27. MaxQuant.org [homepage on the Internet]. Max- Quant. Max planck institute of bio chemistry. Martinsried: Germany; c2013 [cited 2014 January 7]. Available from: / Skyline.gs.washington.edu [homepage on the Internet]. MacCoss Lab Software. Washington: University of Washington, Seattle; c2014 [cited 2014 January 7]. Available from: / skyline.gs.washington.edu/ labkey/ wiki/ home/ software/ Skyline/ page.view?name=defaude. 29. Ingenuity.com [homepage on the Internet]. Ingenuity. CA: Redwood City [cited 2014 January 7]. Available from: / David.abcc.ncifcrf.gov [homepage on the Internet]. DAVID Bioinformatics Resources 6.7. National institute of allergy and infectious diseases, Frederick, MD [cited 2014 January 7]. Available from: / david.abcc.ncifcrf. gov/ home.jsp. 31. van Iersel MP, Kelder T, Pico AR et al. Presenting and exploring bio logical pathways with PathVisio. BMC Bioinformatics 2008; 9: 399. doi: / S120
NPTEL VIDEO COURSE PROTEOMICS PROF. SANJEEVA SRIVASTAVA
 LECTURE-25 Quantitative proteomics: itraq and TMT TRANSCRIPT Welcome to the proteomics course. Today we will talk about quantitative proteomics and discuss about itraq and TMT techniques. The quantitative
LECTURE-25 Quantitative proteomics: itraq and TMT TRANSCRIPT Welcome to the proteomics course. Today we will talk about quantitative proteomics and discuss about itraq and TMT techniques. The quantitative
PC235: 2008 Lecture 5: Quantitation. Arnold Falick
 PC235: 2008 Lecture 5: Quantitation Arnold Falick falickam@berkeley.edu Summary What you will learn from this lecture: There are many methods to perform quantitation using mass spectrometry (any method
PC235: 2008 Lecture 5: Quantitation Arnold Falick falickam@berkeley.edu Summary What you will learn from this lecture: There are many methods to perform quantitation using mass spectrometry (any method
Modeling Mass Spectrometry-Based Protein Analysis
 Chapter 8 Jan Eriksson and David Fenyö Abstract The success of mass spectrometry based proteomics depends on efficient methods for data analysis. These methods require a detailed understanding of the information
Chapter 8 Jan Eriksson and David Fenyö Abstract The success of mass spectrometry based proteomics depends on efficient methods for data analysis. These methods require a detailed understanding of the information
Mass Spectrometry and Proteomics - Lecture 5 - Matthias Trost Newcastle University
 Mass Spectrometry and Proteomics - Lecture 5 - Matthias Trost Newcastle University matthias.trost@ncl.ac.uk Previously Proteomics Sample prep 144 Lecture 5 Quantitation techniques Search Algorithms Proteomics
Mass Spectrometry and Proteomics - Lecture 5 - Matthias Trost Newcastle University matthias.trost@ncl.ac.uk Previously Proteomics Sample prep 144 Lecture 5 Quantitation techniques Search Algorithms Proteomics
Aplicació de la proteòmica a la cerca de Biomarcadors proteics Barcelona, 08 de Juny 2010
 Aplicació de la proteòmica a la cerca de Biomarcadors proteics Barcelona, 8 de Juny 21 Eliandre de Oliveira Plataforma de Proteòmica Parc Científic de Barcelona Protein Chemistry Proteomics Hypothesis-free
Aplicació de la proteòmica a la cerca de Biomarcadors proteics Barcelona, 8 de Juny 21 Eliandre de Oliveira Plataforma de Proteòmica Parc Científic de Barcelona Protein Chemistry Proteomics Hypothesis-free
Quantitative Proteomics
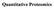 Quantitative Proteomics Quantitation AND Mass Spectrometry Condition A Condition B Identify and quantify differently expressed proteins resulting from a change in the environment (stimulus, disease) Lyse
Quantitative Proteomics Quantitation AND Mass Spectrometry Condition A Condition B Identify and quantify differently expressed proteins resulting from a change in the environment (stimulus, disease) Lyse
Workflow concept. Data goes through the workflow. A Node contains an operation An edge represents data flow The results are brought together in tables
 PROTEOME DISCOVERER Workflow concept Data goes through the workflow Spectra Peptides Quantitation A Node contains an operation An edge represents data flow The results are brought together in tables Protein
PROTEOME DISCOVERER Workflow concept Data goes through the workflow Spectra Peptides Quantitation A Node contains an operation An edge represents data flow The results are brought together in tables Protein
Workshop: SILAC and Alternative Labeling Strategies in Quantitative Proteomics
 Workshop: SILAC and Alternative Labeling Strategies in Quantitative Proteomics SILAC and Stable Isotope Dimethyl-Labeling Approaches in Quantitative Proteomics Ho-Tak Lau, Hyong-Won Suh, Shao-En Ong UW
Workshop: SILAC and Alternative Labeling Strategies in Quantitative Proteomics SILAC and Stable Isotope Dimethyl-Labeling Approaches in Quantitative Proteomics Ho-Tak Lau, Hyong-Won Suh, Shao-En Ong UW
SILAC and TMT. IDeA National Resource for Proteomics Workshop for Graduate Students and Post-docs Renny Lan 5/18/2017
 SILAC and TMT IDeA National Resource for Proteomics Workshop for Graduate Students and Post-docs Renny Lan 5/18/2017 UHPLC peak chosen at 26.47 min LC Mass at 571.36 chosen for MS/MS MS/MS MS This is a
SILAC and TMT IDeA National Resource for Proteomics Workshop for Graduate Students and Post-docs Renny Lan 5/18/2017 UHPLC peak chosen at 26.47 min LC Mass at 571.36 chosen for MS/MS MS/MS MS This is a
Tandem mass spectra were extracted from the Xcalibur data system format. (.RAW) and charge state assignment was performed using in house software
 Supplementary Methods Software Interpretation of Tandem mass spectra Tandem mass spectra were extracted from the Xcalibur data system format (.RAW) and charge state assignment was performed using in house
Supplementary Methods Software Interpretation of Tandem mass spectra Tandem mass spectra were extracted from the Xcalibur data system format (.RAW) and charge state assignment was performed using in house
Key questions of proteomics. Bioinformatics 2. Proteomics. Foundation of proteomics. What proteins are there? Protein digestion
 s s Key questions of proteomics What proteins are there? Bioinformatics 2 Lecture 2 roteomics How much is there of each of the proteins? - Absolute quantitation - Stoichiometry What (modification/splice)
s s Key questions of proteomics What proteins are there? Bioinformatics 2 Lecture 2 roteomics How much is there of each of the proteins? - Absolute quantitation - Stoichiometry What (modification/splice)
Computational Methods for Mass Spectrometry Proteomics
 Computational Methods for Mass Spectrometry Proteomics Eidhammer, Ingvar ISBN-13: 9780470512975 Table of Contents Preface. Acknowledgements. 1 Protein, Proteome, and Proteomics. 1.1 Primary goals for studying
Computational Methods for Mass Spectrometry Proteomics Eidhammer, Ingvar ISBN-13: 9780470512975 Table of Contents Preface. Acknowledgements. 1 Protein, Proteome, and Proteomics. 1.1 Primary goals for studying
Increasing the Multiplexing of Protein Quantitation from 6- to 10-Plex with Reporter Ion Isotopologues
 Increasing the Multiplexing of Protein Quantitation from 6- to 1-Plex with Reporter Ion Isotopologues Rosa Viner, 1 Ryan Bomgarden, 2 Michael Blank, 1 John Rogers 2 1 Thermo Fisher Scientific, San Jose,
Increasing the Multiplexing of Protein Quantitation from 6- to 1-Plex with Reporter Ion Isotopologues Rosa Viner, 1 Ryan Bomgarden, 2 Michael Blank, 1 John Rogers 2 1 Thermo Fisher Scientific, San Jose,
Protein analysis using mass spectrometry
 Protein analysis using mass spectrometry Michael Stadlmeier 2017/12/18 Literature http://www.carellgroup.de/teaching/master 3 What is Proteomics? The proteome is: the entire set of proteins in a given
Protein analysis using mass spectrometry Michael Stadlmeier 2017/12/18 Literature http://www.carellgroup.de/teaching/master 3 What is Proteomics? The proteome is: the entire set of proteins in a given
Isotopic-Labeling and Mass Spectrometry-Based Quantitative Proteomics
 Isotopic-Labeling and Mass Spectrometry-Based Quantitative Proteomics Xiao-jun Li, Ph.D. Current address: Homestead Clinical Day 4 October 19, 2006 Protein Quantification LC-MS/MS Data XLink mzxml file
Isotopic-Labeling and Mass Spectrometry-Based Quantitative Proteomics Xiao-jun Li, Ph.D. Current address: Homestead Clinical Day 4 October 19, 2006 Protein Quantification LC-MS/MS Data XLink mzxml file
Advances in quantitative proteomics using stable isotope tags
 Advances in quantitative proteomics using stable isotope tags Mark R. Flory, Timothy J. Griffin, Daniel Martin and Ruedi Aebersold A great deal of current biological and clinical research is directed at
Advances in quantitative proteomics using stable isotope tags Mark R. Flory, Timothy J. Griffin, Daniel Martin and Ruedi Aebersold A great deal of current biological and clinical research is directed at
Research Article High Mass Accuracy Phosphopeptide Identification Using Tandem Mass Spectra
 Proteomics Volume 2012, Article ID 104681, 5 pages doi:10.1155/2012/104681 Research Article High Mass Accuracy Phosphopeptide Identification Using Tandem Mass Spectra Rovshan G. Sadygov Sealy Center for
Proteomics Volume 2012, Article ID 104681, 5 pages doi:10.1155/2012/104681 Research Article High Mass Accuracy Phosphopeptide Identification Using Tandem Mass Spectra Rovshan G. Sadygov Sealy Center for
Quantitation of TMT-Labeled Peptides Using Higher-Energy Collisional Dissociation on the Velos Pro Ion Trap Mass Spectrometer
 Application Note: 520 Quantitation of TMT-Labeled Peptides Using Higher-Energy Collisional Dissociation on the Velos Pro Ion Trap Mass Spectrometer Roger G. Biringer, Julie A. Horner, Rosa Viner, Andreas
Application Note: 520 Quantitation of TMT-Labeled Peptides Using Higher-Energy Collisional Dissociation on the Velos Pro Ion Trap Mass Spectrometer Roger G. Biringer, Julie A. Horner, Rosa Viner, Andreas
A label-free quantification method by MS/MS TIC compared to SILAC and spectral counting in a proteomics screen
 994 DOI 10.1002/pmic.200700426 Proteomics 2008, 8, 994 999 TECHNICAL BRIEF A label-free quantification method by MS/MS TIC compared to SILAC and spectral counting in a proteomics screen John M. Asara 1,
994 DOI 10.1002/pmic.200700426 Proteomics 2008, 8, 994 999 TECHNICAL BRIEF A label-free quantification method by MS/MS TIC compared to SILAC and spectral counting in a proteomics screen John M. Asara 1,
Methods for proteome analysis of obesity (Adipose tissue)
 Methods for proteome analysis of obesity (Adipose tissue) I. Sample preparation and liquid chromatography-tandem mass spectrometric analysis Instruments, softwares, and materials AB SCIEX Triple TOF 5600
Methods for proteome analysis of obesity (Adipose tissue) I. Sample preparation and liquid chromatography-tandem mass spectrometric analysis Instruments, softwares, and materials AB SCIEX Triple TOF 5600
Quantitation of a target protein in crude samples using targeted peptide quantification by Mass Spectrometry
 Quantitation of a target protein in crude samples using targeted peptide quantification by Mass Spectrometry Jon Hao, Rong Ye, and Mason Tao Poochon Scientific, Frederick, Maryland 21701 Abstract Background:
Quantitation of a target protein in crude samples using targeted peptide quantification by Mass Spectrometry Jon Hao, Rong Ye, and Mason Tao Poochon Scientific, Frederick, Maryland 21701 Abstract Background:
Chapter 4. strategies for protein quantitation Ⅱ
 Proteomics Chapter 4. strategies for protein quantitation Ⅱ 1 Multiplexed proteomics Multiplexed proteomics is the use of fluorescent stains or probes with different excitation and emission spectra to
Proteomics Chapter 4. strategies for protein quantitation Ⅱ 1 Multiplexed proteomics Multiplexed proteomics is the use of fluorescent stains or probes with different excitation and emission spectra to
Protein Sequencing Research Group ABRF 2015 annual meeting
 Protein Sequencing Research Group ABRF 2015 annual meeting » N-terminal sequencing is in the midst of a technology transition from classical Edman sequencing to mass spectrometry (MS)-based sequencing»
Protein Sequencing Research Group ABRF 2015 annual meeting » N-terminal sequencing is in the midst of a technology transition from classical Edman sequencing to mass spectrometry (MS)-based sequencing»
Mass spectrometry-based proteomics has become
 FOCUS: THE ORBITRAP Computational Principles of Determining and Improving Mass Precision and Accuracy for Proteome Measurements in an Orbitrap Jürgen Cox and Matthias Mann Proteomics and Signal Transduction,
FOCUS: THE ORBITRAP Computational Principles of Determining and Improving Mass Precision and Accuracy for Proteome Measurements in an Orbitrap Jürgen Cox and Matthias Mann Proteomics and Signal Transduction,
Comprehensive support for quantitation
 Comprehensive support for quantitation One of the major new features in the current release of Mascot is support for quantitation. This is still work in progress. Our goal is to support all of the popular
Comprehensive support for quantitation One of the major new features in the current release of Mascot is support for quantitation. This is still work in progress. Our goal is to support all of the popular
Peptide Labeling with Isobaric Tags Yields Higher Identification Rates Using itraq 4-Plex Compared to TMT 6-Plex and itraq 8-Plex on LTQ Orbitrap
 Anal. Chem. 2010, 82, 6549 6558 Peptide Labeling with Isobaric Tags Yields Higher Identification Rates Using itraq 4-Plex Compared to TMT 6-Plex and itraq 8-Plex on LTQ Orbitrap Peter Pichler,*, Thomas
Anal. Chem. 2010, 82, 6549 6558 Peptide Labeling with Isobaric Tags Yields Higher Identification Rates Using itraq 4-Plex Compared to TMT 6-Plex and itraq 8-Plex on LTQ Orbitrap Peter Pichler,*, Thomas
Towards the Prediction of Protein Abundance from Tandem Mass Spectrometry Data
 Towards the Prediction of Protein Abundance from Tandem Mass Spectrometry Data Anthony J Bonner Han Liu Abstract This paper addresses a central problem of Proteomics: estimating the amounts of each of
Towards the Prediction of Protein Abundance from Tandem Mass Spectrometry Data Anthony J Bonner Han Liu Abstract This paper addresses a central problem of Proteomics: estimating the amounts of each of
We are IntechOpen, the first native scientific publisher of Open Access books. International authors and editors. Our authors are among the TOP 1%
 We are IntechOpen, the first native scientific publisher of Open Access books 3,350 108,000 1.7 M Open access books available International authors and editors Downloads Our authors are among the 151 Countries
We are IntechOpen, the first native scientific publisher of Open Access books 3,350 108,000 1.7 M Open access books available International authors and editors Downloads Our authors are among the 151 Countries
Qualitative Proteomics (how to obtain high-confidence high-throughput protein identification!)
 Qualitative Proteomics (how to obtain high-confidence high-throughput protein identification!) James A. Mobley, Ph.D. Director of Research in Urology Associate Director of Mass Spectrometry (contact: mobleyja@uab.edu)
Qualitative Proteomics (how to obtain high-confidence high-throughput protein identification!) James A. Mobley, Ph.D. Director of Research in Urology Associate Director of Mass Spectrometry (contact: mobleyja@uab.edu)
Protein Quantitation II: Multiple Reaction Monitoring. Kelly Ruggles New York University
 Protein Quantitation II: Multiple Reaction Monitoring Kelly Ruggles kelly@fenyolab.org New York University Traditional Affinity-based proteomics Use antibodies to quantify proteins Western Blot RPPA Immunohistochemistry
Protein Quantitation II: Multiple Reaction Monitoring Kelly Ruggles kelly@fenyolab.org New York University Traditional Affinity-based proteomics Use antibodies to quantify proteins Western Blot RPPA Immunohistochemistry
Amine specific Labeling Reagents for Multiplexed Relative and Absolute Protein Quantitation
 Product Bulletin itraq Reagents itraq Reagents Amine specific Labeling Reagents for Multiplexed Relative and Absolute Protein Quantitation Background Proteomics research includes the characterization of
Product Bulletin itraq Reagents itraq Reagents Amine specific Labeling Reagents for Multiplexed Relative and Absolute Protein Quantitation Background Proteomics research includes the characterization of
Introduction to Proteomics & Bottom-up Proteomics
 Used for MS Short Course at Tsinghua by R. Graham Cooks, Hao Chen, Zheng Ouyang, Andy Tao, Yu Xia and Lingjun Li Introduction to Proteomics & Bottom-up Proteomics W. Andy Tao Purdue University watao@purdue.edu
Used for MS Short Course at Tsinghua by R. Graham Cooks, Hao Chen, Zheng Ouyang, Andy Tao, Yu Xia and Lingjun Li Introduction to Proteomics & Bottom-up Proteomics W. Andy Tao Purdue University watao@purdue.edu
Mass Spectrometry. Hyphenated Techniques GC-MS LC-MS and MS-MS
 Mass Spectrometry Hyphenated Techniques GC-MS LC-MS and MS-MS Reasons for Using Chromatography with MS Mixture analysis by MS alone is difficult Fragmentation from ionization (EI or CI) Fragments from
Mass Spectrometry Hyphenated Techniques GC-MS LC-MS and MS-MS Reasons for Using Chromatography with MS Mixture analysis by MS alone is difficult Fragmentation from ionization (EI or CI) Fragments from
UCD Conway Institute of Biomolecular & Biomedical Research Graduate Education 2009/2010
 EMERGING PROTEOMIC TECHNOLOGIES - MODULE SCHEDULE & OUTLINE 2010 Course Organiser: Dr. Giuliano Elia Module Co-ordinator: Dr Giuliano Elia Credits: 5 Date & Time Session & Topic Coordinator 14th April
EMERGING PROTEOMIC TECHNOLOGIES - MODULE SCHEDULE & OUTLINE 2010 Course Organiser: Dr. Giuliano Elia Module Co-ordinator: Dr Giuliano Elia Credits: 5 Date & Time Session & Topic Coordinator 14th April
Chapter 2 What are the Common Mass Spectrometry-Based Analyses Used in Biology?
 Chapter 2 What are the Common Mass Spectrometry-Based Analyses Used in Biology? Abstract Mass spectrometry is used in many field of research, such as biology, chemistry, geology, etc. The focus of this
Chapter 2 What are the Common Mass Spectrometry-Based Analyses Used in Biology? Abstract Mass spectrometry is used in many field of research, such as biology, chemistry, geology, etc. The focus of this
Mass spectrometry based proteomics
 Practice-oriented, student-friendly modernization of the biomedical education for strengthening the international competitiveness of the rural Hungarian universities TÁMOP-4.1.1.C-13/1/KONV-2014-0001 Mass
Practice-oriented, student-friendly modernization of the biomedical education for strengthening the international competitiveness of the rural Hungarian universities TÁMOP-4.1.1.C-13/1/KONV-2014-0001 Mass
for the Novice Mass Spectrometry (^>, John Greaves and John Roboz yc**' CRC Press J Taylor & Francis Group Boca Raton London New York
 Mass Spectrometry for the Novice John Greaves and John Roboz (^>, yc**' CRC Press J Taylor & Francis Group Boca Raton London New York CRC Press is an imprint of the Taylor & Francis Croup, an informa business
Mass Spectrometry for the Novice John Greaves and John Roboz (^>, yc**' CRC Press J Taylor & Francis Group Boca Raton London New York CRC Press is an imprint of the Taylor & Francis Croup, an informa business
Protein Quantitation II: Multiple Reaction Monitoring. Kelly Ruggles New York University
 Protein Quantitation II: Multiple Reaction Monitoring Kelly Ruggles kelly@fenyolab.org New York University Traditional Affinity-based proteomics Use antibodies to quantify proteins Western Blot Immunohistochemistry
Protein Quantitation II: Multiple Reaction Monitoring Kelly Ruggles kelly@fenyolab.org New York University Traditional Affinity-based proteomics Use antibodies to quantify proteins Western Blot Immunohistochemistry
TANDEM MASS SPECTROSCOPY
 TANDEM MASS SPECTROSCOPY 1 MASS SPECTROMETER TYPES OF MASS SPECTROMETER PRINCIPLE TANDEM MASS SPECTROMETER INSTRUMENTATION QUADRAPOLE MASS ANALYZER TRIPLE QUADRAPOLE MASS ANALYZER TIME OF FLIGHT MASS ANALYSER
TANDEM MASS SPECTROSCOPY 1 MASS SPECTROMETER TYPES OF MASS SPECTROMETER PRINCIPLE TANDEM MASS SPECTROMETER INSTRUMENTATION QUADRAPOLE MASS ANALYZER TRIPLE QUADRAPOLE MASS ANALYZER TIME OF FLIGHT MASS ANALYSER
Identification of Human Hemoglobin Protein Variants Using Electrospray Ionization-Electron Transfer Dissociation Mass Spectrometry
 Identification of Human Hemoglobin Protein Variants Using Electrospray Ionization-Electron Transfer Dissociation Mass Spectrometry Jonathan Williams Waters Corporation, Milford, MA, USA A P P L I C AT
Identification of Human Hemoglobin Protein Variants Using Electrospray Ionization-Electron Transfer Dissociation Mass Spectrometry Jonathan Williams Waters Corporation, Milford, MA, USA A P P L I C AT
MS-based proteomics to investigate proteins and their modifications
 MS-based proteomics to investigate proteins and their modifications Francis Impens VIB Proteomics Core October th 217 Overview Mass spectrometry-based proteomics: general workflow Identification of protein
MS-based proteomics to investigate proteins and their modifications Francis Impens VIB Proteomics Core October th 217 Overview Mass spectrometry-based proteomics: general workflow Identification of protein
Ing. Tomasz Kanik. doc. RNDr. Štefan Peško, CSc.
 Ing. Tomasz Kanik Školiteľ: doc. RNDr. Štefan Peško, CSc. Pracovisko: Študijný program: KMMOA, FRI, ŽU 9.2.9 Aplikovaná informatika 1 identifikácia problémovej skupiny pacientov, zlepšenie kvality rozhodovacích
Ing. Tomasz Kanik Školiteľ: doc. RNDr. Štefan Peško, CSc. Pracovisko: Študijný program: KMMOA, FRI, ŽU 9.2.9 Aplikovaná informatika 1 identifikácia problémovej skupiny pacientov, zlepšenie kvality rozhodovacích
Biological Mass Spectrometry
 Biochemistry 412 Biological Mass Spectrometry February 13 th, 2007 Proteomics The study of the complete complement of proteins found in an organism Degrees of Freedom for Protein Variability Covalent Modifications
Biochemistry 412 Biological Mass Spectrometry February 13 th, 2007 Proteomics The study of the complete complement of proteins found in an organism Degrees of Freedom for Protein Variability Covalent Modifications
Mass spectrometry and proteomics Steven P Gygi* and Ruedi Aebersold
 489 Mass spectrometry and proteomics Steven P Gygi* and Ruedi Aebersold Proteomics is the systematic analysis of the proteins expressed by a cell or tissue, and mass spectrometry is its essential analytical
489 Mass spectrometry and proteomics Steven P Gygi* and Ruedi Aebersold Proteomics is the systematic analysis of the proteins expressed by a cell or tissue, and mass spectrometry is its essential analytical
Improved Precision of itraq and TMT Quantification by an Axial Extraction Field in an Orbitrap HCD Cell
 pubs.acs.org/ac Improved Precision of itraq and TMT Quantification by an Axial Extraction Field in an Orbitrap HCD Cell Peter Pichler,*, Thomas K ocher, Johann Holzmann, Thomas M ohring, Gustav Ammerer,,
pubs.acs.org/ac Improved Precision of itraq and TMT Quantification by an Axial Extraction Field in an Orbitrap HCD Cell Peter Pichler,*, Thomas K ocher, Johann Holzmann, Thomas M ohring, Gustav Ammerer,,
Designed for Accuracy. Innovation with Integrity. High resolution quantitative proteomics LC-MS
 Designed for Accuracy High resolution quantitative proteomics Innovation with Integrity LC-MS Setting New Standards in Accuracy The development of mass spectrometry based proteomics approaches has dramatically
Designed for Accuracy High resolution quantitative proteomics Innovation with Integrity LC-MS Setting New Standards in Accuracy The development of mass spectrometry based proteomics approaches has dramatically
A Quadrupole-Orbitrap Hybrid Mass Spectrometer Offers Highest Benchtop Performance for In-Depth Analysis of Complex Proteomes
 A Quadrupole-Orbitrap Hybrid Mass Spectrometer Offers Highest Benchtop Performance for In-Depth Analysis of Complex Proteomes Zhiqi Hao 1, Yi Zhang 1, Shannon Eliuk 1, Justin Blethrow 1, Dave Horn 1, Vlad
A Quadrupole-Orbitrap Hybrid Mass Spectrometer Offers Highest Benchtop Performance for In-Depth Analysis of Complex Proteomes Zhiqi Hao 1, Yi Zhang 1, Shannon Eliuk 1, Justin Blethrow 1, Dave Horn 1, Vlad
Yifei Bao. Beatrix. Manor Askenazi
 Detection and Correction of Interference in MS1 Quantitation of Peptides Using their Isotope Distributions Yifei Bao Department of Computer Science Stevens Institute of Technology Beatrix Ueberheide Department
Detection and Correction of Interference in MS1 Quantitation of Peptides Using their Isotope Distributions Yifei Bao Department of Computer Science Stevens Institute of Technology Beatrix Ueberheide Department
Protein Catalog Number Quantity Myoglobin Sigma M ug
 Dear Colleagues, November 6, 2014 Please find enclosed the ABRF 2015 PSRG samples that you requested from the ABRF Protein Sequence Research Group. This is the 27 th study in an annual series designed
Dear Colleagues, November 6, 2014 Please find enclosed the ABRF 2015 PSRG samples that you requested from the ABRF Protein Sequence Research Group. This is the 27 th study in an annual series designed
PROTEOMICS IN VASCULAR BIOLOGY
 ESC Summerschool 2013 Nice ESC Summer School Cardiovascular Sciences June 18, 2013 PROTEOMICS IN VASCULAR BIOLOGY Prof dr Anton J.G. Horrevoets Molecular Cell Biology and Immunology VU Medical Center Amsterdam
ESC Summerschool 2013 Nice ESC Summer School Cardiovascular Sciences June 18, 2013 PROTEOMICS IN VASCULAR BIOLOGY Prof dr Anton J.G. Horrevoets Molecular Cell Biology and Immunology VU Medical Center Amsterdam
Atomic masses. Atomic masses of elements. Atomic masses of isotopes. Nominal and exact atomic masses. Example: CO, N 2 ja C 2 H 4
 High-Resolution Mass spectrometry (HR-MS, HRAM-MS) (FT mass spectrometry) MS that enables identifying elemental compositions (empirical formulas) from accurate m/z data 9.05.2017 1 Atomic masses (atomic
High-Resolution Mass spectrometry (HR-MS, HRAM-MS) (FT mass spectrometry) MS that enables identifying elemental compositions (empirical formulas) from accurate m/z data 9.05.2017 1 Atomic masses (atomic
Quantitative analysis of the proteome
 BMG 744 Proteomics-Mass Spectrometry Quantitative analysis of the proteome Stephen Barnes, PhD sbarnes@uab.edu 1 The ASMS 1996-2007 www.tagate.com/western/wild_west.jpg 2 1 Proteomics Data Standards 2005
BMG 744 Proteomics-Mass Spectrometry Quantitative analysis of the proteome Stephen Barnes, PhD sbarnes@uab.edu 1 The ASMS 1996-2007 www.tagate.com/western/wild_west.jpg 2 1 Proteomics Data Standards 2005
Overview - MS Proteomics in One Slide. MS masses of peptides. MS/MS fragments of a peptide. Results! Match to sequence database
 Overview - MS Proteomics in One Slide Obtain protein Digest into peptides Acquire spectra in mass spectrometer MS masses of peptides MS/MS fragments of a peptide Results! Match to sequence database 2 But
Overview - MS Proteomics in One Slide Obtain protein Digest into peptides Acquire spectra in mass spectrometer MS masses of peptides MS/MS fragments of a peptide Results! Match to sequence database 2 But
Combining low- and high-energy tandem mass spectra for optimized peptide quantification with isobaric tags
 available at www.sciencedirect.com www.elsevier.com/locate/jprot Combining low- and high-energy tandem mass spectra for optimized peptide quantification with isobaric tags Loïc Dayon a, Carla Pasquarello
available at www.sciencedirect.com www.elsevier.com/locate/jprot Combining low- and high-energy tandem mass spectra for optimized peptide quantification with isobaric tags Loïc Dayon a, Carla Pasquarello
Quantitative Proteomics
 BSPR workshop 16 th July 2010 Quantitative Proteomics Kathryn Lilley Cambridge Centre for Proteomics Department of Biochemistry University of Cambridge k.s.lilley@bioc.cam.ac.uk www.bio.cam.ac.uk/proteomics/
BSPR workshop 16 th July 2010 Quantitative Proteomics Kathryn Lilley Cambridge Centre for Proteomics Department of Biochemistry University of Cambridge k.s.lilley@bioc.cam.ac.uk www.bio.cam.ac.uk/proteomics/
Optimization and Use of Peptide Mass Measurement Accuracy in Shotgun Proteomics* S
 Research Optimization and Use of Peptide Mass Measurement Accuracy in Shotgun Proteomics* S Wilhelm Haas, Brendan K. Faherty, Scott A. Gerber, Joshua E. Elias, Sean A. Beausoleil, Corey E. Bakalarski,
Research Optimization and Use of Peptide Mass Measurement Accuracy in Shotgun Proteomics* S Wilhelm Haas, Brendan K. Faherty, Scott A. Gerber, Joshua E. Elias, Sean A. Beausoleil, Corey E. Bakalarski,
High-Field Orbitrap Creating new possibilities
 Thermo Scientific Orbitrap Elite Hybrid Mass Spectrometer High-Field Orbitrap Creating new possibilities Ultrahigh resolution Faster scanning Higher sensitivity Complementary fragmentation The highest
Thermo Scientific Orbitrap Elite Hybrid Mass Spectrometer High-Field Orbitrap Creating new possibilities Ultrahigh resolution Faster scanning Higher sensitivity Complementary fragmentation The highest
Kapitola S5. Skrutkovica na rotačnej ploche
 Kapitola S5 Skrutkovica na rotačnej ploche Nech je rotačná plocha určená osou rotácie o a meridiánom m. Skrutkový pohyb je pohyb zložený z rovnomerného rotačného pohybu okolo osi o a z rovnomerného translačného
Kapitola S5 Skrutkovica na rotačnej ploche Nech je rotačná plocha určená osou rotácie o a meridiánom m. Skrutkový pohyb je pohyb zložený z rovnomerného rotačného pohybu okolo osi o a z rovnomerného translačného
Proteome-wide label-free quantification with MaxQuant. Jürgen Cox Max Planck Institute of Biochemistry July 2011
 Proteome-wide label-free quantification with MaxQuant Jürgen Cox Max Planck Institute of Biochemistry July 2011 MaxQuant MaxQuant Feature detection Data acquisition Initial Andromeda search Statistics
Proteome-wide label-free quantification with MaxQuant Jürgen Cox Max Planck Institute of Biochemistry July 2011 MaxQuant MaxQuant Feature detection Data acquisition Initial Andromeda search Statistics
Improved 6- Plex TMT Quantification Throughput Using a Linear Ion Trap HCD MS 3 Scan Jane M. Liu, 1,2 * Michael J. Sweredoski, 2 Sonja Hess 2 *
 Improved 6- Plex TMT Quantification Throughput Using a Linear Ion Trap HCD MS 3 Scan Jane M. Liu, 1,2 * Michael J. Sweredoski, 2 Sonja Hess 2 * 1 Department of Chemistry, Pomona College, Claremont, California
Improved 6- Plex TMT Quantification Throughput Using a Linear Ion Trap HCD MS 3 Scan Jane M. Liu, 1,2 * Michael J. Sweredoski, 2 Sonja Hess 2 * 1 Department of Chemistry, Pomona College, Claremont, California
Targeted Proteomics Environment
 Targeted Proteomics Environment Quantitative Proteomics with Bruker Q-TOF Instruments and Skyline Brendan MacLean Quantitative Proteomics Spectrum-based Spectral counting Isobaric tags Chromatography-based
Targeted Proteomics Environment Quantitative Proteomics with Bruker Q-TOF Instruments and Skyline Brendan MacLean Quantitative Proteomics Spectrum-based Spectral counting Isobaric tags Chromatography-based
Isobaric Labeling-Based Relative Quantification in Shotgun Proteomics
 This is an open access article published under an ACS AuthorChoice License, which permits copying and redistribution of the article or any adaptations for non-commercial purposes. pubs.acs.org/jpr Isobaric
This is an open access article published under an ACS AuthorChoice License, which permits copying and redistribution of the article or any adaptations for non-commercial purposes. pubs.acs.org/jpr Isobaric
Chem 250 Unit 1 Proteomics by Mass Spectrometry
 Chem 250 Unit 1 Proteomics by Mass Spectrometry Article #1 Quantitative MS for proteomics: teaching a new dog old tricks. MacCoss MJ, Matthews DE., Anal Chem. 2005 Aug 1;77(15):294A-302A. 1. Synopsis 1.1.
Chem 250 Unit 1 Proteomics by Mass Spectrometry Article #1 Quantitative MS for proteomics: teaching a new dog old tricks. MacCoss MJ, Matthews DE., Anal Chem. 2005 Aug 1;77(15):294A-302A. 1. Synopsis 1.1.
See for options on how to legitimately share published articles.
 Downloaded via 148.251.232.83 on December 26, 2018 at 05:35:30 (UTC). See https://pubs.acs.org/sharingguidelines for options on how to legitimately share published articles. 294 NLYTICL CHEMISTRY / UGUST
Downloaded via 148.251.232.83 on December 26, 2018 at 05:35:30 (UTC). See https://pubs.acs.org/sharingguidelines for options on how to legitimately share published articles. 294 NLYTICL CHEMISTRY / UGUST
PROTEIN SEQUENCING AND IDENTIFICATION USING TANDEM MASS SPECTROMETRY
 PROTEIN SEQUENCING AND IDENTIFICATION USING TANDEM MASS SPECTROMETRY Michael Kinter Department of Cell Biology Lerner Research Institute Cleveland Clinic Foundation Nicholas E. Sherman Department of Microbiology
PROTEIN SEQUENCING AND IDENTIFICATION USING TANDEM MASS SPECTROMETRY Michael Kinter Department of Cell Biology Lerner Research Institute Cleveland Clinic Foundation Nicholas E. Sherman Department of Microbiology
Pro tein quantification has become an important. Peptide and Protein Quantification Using itraq with Electron Transfer Dissociation
 Peptide and Protein Quantification Using itraq with Electron Transfer Dissociation Doug Phanstiel," Yi Zhang," [arrod A. Marte,'>' and Joshua J. Coon'':" a Department of Chemistry, University of Wisconsin,
Peptide and Protein Quantification Using itraq with Electron Transfer Dissociation Doug Phanstiel," Yi Zhang," [arrod A. Marte,'>' and Joshua J. Coon'':" a Department of Chemistry, University of Wisconsin,
Technical Note. Introduction
 Technical Note Analysis and Characterization of Psilocybin and Psilocin Using Liquid Chromatography - Electrospray Ionization Mass Spectrometry (LC-ESI-MS) with Collision-Induced-Dissociation (CID) and
Technical Note Analysis and Characterization of Psilocybin and Psilocin Using Liquid Chromatography - Electrospray Ionization Mass Spectrometry (LC-ESI-MS) with Collision-Induced-Dissociation (CID) and
Peter A. DiMaggio, Jr., Nicolas L. Young, Richard C. Baliban, Benjamin A. Garcia, and Christodoulos A. Floudas. Research
 Research A Mixed Integer Linear Optimization Framework for the Identification and Quantification of Targeted Post-translational Modifications of Highly Modified Proteins Using Multiplexed Electron Transfer
Research A Mixed Integer Linear Optimization Framework for the Identification and Quantification of Targeted Post-translational Modifications of Highly Modified Proteins Using Multiplexed Electron Transfer
Relative quantification using TMT11plex on a modified Q Exactive HF mass spectrometer
 POSTER NOTE 6558 Relative quantification using TMT11plex on a modified mass spectrometer Authors Tabiwang N. Arrey, 1 Rosa Viner, 2 Ryan D. Bomgarden, 3 Eugen Damoc, 1 Markus Kellmann, 1 Thomas Moehring,
POSTER NOTE 6558 Relative quantification using TMT11plex on a modified mass spectrometer Authors Tabiwang N. Arrey, 1 Rosa Viner, 2 Ryan D. Bomgarden, 3 Eugen Damoc, 1 Markus Kellmann, 1 Thomas Moehring,
Statistical analysis of isobaric-labeled mass spectrometry data
 Statistical analysis of isobaric-labeled mass spectrometry data Farhad Shakeri July 3, 2018 Core Unit for Bioinformatics Analyses Institute for Genomic Statistics and Bioinformatics University Hospital
Statistical analysis of isobaric-labeled mass spectrometry data Farhad Shakeri July 3, 2018 Core Unit for Bioinformatics Analyses Institute for Genomic Statistics and Bioinformatics University Hospital
Introduction to Mass Spectrometry and Proteomics
 Introduction to Mass Spectrometry and Proteomics Annie Moradian Proteome Exploration Laboratory Contents 1. Basic Principles of Mass Spectrometry Ionization Sources Mass Analyzers Detectors 2. Tandem Mass
Introduction to Mass Spectrometry and Proteomics Annie Moradian Proteome Exploration Laboratory Contents 1. Basic Principles of Mass Spectrometry Ionization Sources Mass Analyzers Detectors 2. Tandem Mass
Optimization and Use of Peptide Mass Measurement Accuracy in Shotgun Proteomics
 MCP Papers in Press. Published on April 23, 26 as Manuscript M5339-MCP2 Optimization and Use of Peptide Mass Measurement Accuracy in Shotgun Proteomics Wilhelm Haas, Brendan K. Faherty, Scott A. Gerber,
MCP Papers in Press. Published on April 23, 26 as Manuscript M5339-MCP2 Optimization and Use of Peptide Mass Measurement Accuracy in Shotgun Proteomics Wilhelm Haas, Brendan K. Faherty, Scott A. Gerber,
Chromatographic Methods: Basics, Advanced HPLC Methods
 Chromatographic Methods: Basics, Advanced HPLC Methods Hendrik Küpper, Advanced Course on Bioinorganic Chemistry & Biophysics of Plants, summer semester 2018 Chromatography: Basics Chromatography a physical
Chromatographic Methods: Basics, Advanced HPLC Methods Hendrik Küpper, Advanced Course on Bioinorganic Chemistry & Biophysics of Plants, summer semester 2018 Chromatography: Basics Chromatography a physical
Mass Spectrometry. Electron Ionization and Chemical Ionization
 Mass Spectrometry Electron Ionization and Chemical Ionization Mass Spectrometer All Instruments Have: 1. Sample Inlet 2. Ion Source 3. Mass Analyzer 4. Detector 5. Data System http://www.asms.org Ionization
Mass Spectrometry Electron Ionization and Chemical Ionization Mass Spectrometer All Instruments Have: 1. Sample Inlet 2. Ion Source 3. Mass Analyzer 4. Detector 5. Data System http://www.asms.org Ionization
Reagents. Affinity Tag (Biotin) Acid Cleavage Site. Figure 1. Cleavable ICAT Reagent Structure.
 DATA SHEET Protein Expression Analysis Reagents Background The ultimate goal of proteomics is to identify and quantify proteins that are relevant to a given biological state; and to unearth networks of
DATA SHEET Protein Expression Analysis Reagents Background The ultimate goal of proteomics is to identify and quantify proteins that are relevant to a given biological state; and to unearth networks of
Analysis of chemically modified proteins by FT-ICR MS
 Analysis of chemically modified proteins by FT-ICR MS PETR NOVAK EU FT-ICR MS End User School 1 UEF Chemistry August 19-24 2018 Joensuu, Finland Structural Mass Spectrometry Disulfide bonds mapping Limited
Analysis of chemically modified proteins by FT-ICR MS PETR NOVAK EU FT-ICR MS End User School 1 UEF Chemistry August 19-24 2018 Joensuu, Finland Structural Mass Spectrometry Disulfide bonds mapping Limited
HOWTO, example workflow and data files. (Version )
 HOWTO, example workflow and data files. (Version 20 09 2017) 1 Introduction: SugarQb is a collection of software tools (Nodes) which enable the automated identification of intact glycopeptides from HCD
HOWTO, example workflow and data files. (Version 20 09 2017) 1 Introduction: SugarQb is a collection of software tools (Nodes) which enable the automated identification of intact glycopeptides from HCD
SERVA ICPL Kit (Cat.-No )
 INSTRUCTION MANUAL SERVA ICPL Kit (Cat.-No. 39230.01) SERVA Electrophoresis GmbH Carl-Benz-Str. 7 D-69115 Heidelberg Phone +49-6221-138400, Fax +49-6221-1384010 e-mail: info@serva.de http://www.serva.de
INSTRUCTION MANUAL SERVA ICPL Kit (Cat.-No. 39230.01) SERVA Electrophoresis GmbH Carl-Benz-Str. 7 D-69115 Heidelberg Phone +49-6221-138400, Fax +49-6221-1384010 e-mail: info@serva.de http://www.serva.de
(Bio)chemical Proteomics. Alex Kentsis October, 2013
 (Bio)chemical Proteomics Alex Kentsis October, 2013 http://alexkentsis.net A brief history of chemical proteomics 1907: Eduard Buchner, demonstration of cell-free alcohol fermentation (i.e. enzymes) 1946:
(Bio)chemical Proteomics Alex Kentsis October, 2013 http://alexkentsis.net A brief history of chemical proteomics 1907: Eduard Buchner, demonstration of cell-free alcohol fermentation (i.e. enzymes) 1946:
Analysis of Peptide MS/MS Spectra from Large-Scale Proteomics Experiments Using Spectrum Libraries
 Anal. Chem. 2006, 78, 5678-5684 Analysis of Peptide MS/MS Spectra from Large-Scale Proteomics Experiments Using Spectrum Libraries Barbara E. Frewen, Gennifer E. Merrihew, Christine C. Wu, William Stafford
Anal. Chem. 2006, 78, 5678-5684 Analysis of Peptide MS/MS Spectra from Large-Scale Proteomics Experiments Using Spectrum Libraries Barbara E. Frewen, Gennifer E. Merrihew, Christine C. Wu, William Stafford
BIOINF 4120 Bioinformatics 2 - Structures and Systems - Oliver Kohlbacher Summer Systems Biology Exp. Methods
 BIOINF 4120 Bioinformatics 2 - Structures and Systems - Oliver Kohlbacher Summer 2013 14. Systems Biology Exp. Methods Overview Transcriptomics Basics of microarrays Comparative analysis Interactomics:
BIOINF 4120 Bioinformatics 2 - Structures and Systems - Oliver Kohlbacher Summer 2013 14. Systems Biology Exp. Methods Overview Transcriptomics Basics of microarrays Comparative analysis Interactomics:
Quantitative analysis of the proteome. Proteomics Data Standards
 BMG 744 Proteomics-Mass Spectrometry Quantitative analysis of the proteome Stephen Barnes, PhD sbarnes@uab.edu 1 Proteomics Data Standards 2005 MCP Paris guidelines 2008 HUPO MIAPE(Minimum Information
BMG 744 Proteomics-Mass Spectrometry Quantitative analysis of the proteome Stephen Barnes, PhD sbarnes@uab.edu 1 Proteomics Data Standards 2005 MCP Paris guidelines 2008 HUPO MIAPE(Minimum Information
Mass spectrometry has been used a lot in biology since the late 1950 s. However it really came into play in the late 1980 s once methods were
 Mass spectrometry has been used a lot in biology since the late 1950 s. However it really came into play in the late 1980 s once methods were developed to allow the analysis of large intact (bigger than
Mass spectrometry has been used a lot in biology since the late 1950 s. However it really came into play in the late 1980 s once methods were developed to allow the analysis of large intact (bigger than
BST 226 Statistical Methods for Bioinformatics David M. Rocke. January 22, 2014 BST 226 Statistical Methods for Bioinformatics 1
 BST 226 Statistical Methods for Bioinformatics David M. Rocke January 22, 2014 BST 226 Statistical Methods for Bioinformatics 1 Mass Spectrometry Mass spectrometry (mass spec, MS) comprises a set of instrumental
BST 226 Statistical Methods for Bioinformatics David M. Rocke January 22, 2014 BST 226 Statistical Methods for Bioinformatics 1 Mass Spectrometry Mass spectrometry (mass spec, MS) comprises a set of instrumental
Quan%ta%on with XPRESS. and. ASAPRa%o
 Quan%ta%on with XPRESS and ASAPRa%o 1 Pep%de and Protein Quan%ta%on Raw Mass Spec Data Pep%de Iden%fica%on Pep%de Valida%on Quan%ta%on Protein Assignment Protein List msconvert X!Tandem SpectraST SEQUEST*
Quan%ta%on with XPRESS and ASAPRa%o 1 Pep%de and Protein Quan%ta%on Raw Mass Spec Data Pep%de Iden%fica%on Pep%de Valida%on Quan%ta%on Protein Assignment Protein List msconvert X!Tandem SpectraST SEQUEST*
Hydrophilic Interaction Liquid Chromatography: Some Aspects of Solvent and Column Selectivity
 Hydrophilic Interaction Liquid Chromatography: Some Aspects of Solvent and Column Selectivity Monica Dolci, Thermo Fisher Scientific, Runcorn, Cheshire, UK Technical Note 20544 Key Words Hydrophilic, HILIC,
Hydrophilic Interaction Liquid Chromatography: Some Aspects of Solvent and Column Selectivity Monica Dolci, Thermo Fisher Scientific, Runcorn, Cheshire, UK Technical Note 20544 Key Words Hydrophilic, HILIC,
Identification of proteins by enzyme digestion, mass
 Method for Screening Peptide Fragment Ion Mass Spectra Prior to Database Searching Roger E. Moore, Mary K. Young, and Terry D. Lee Beckman Research Institute of the City of Hope, Duarte, California, USA
Method for Screening Peptide Fragment Ion Mass Spectra Prior to Database Searching Roger E. Moore, Mary K. Young, and Terry D. Lee Beckman Research Institute of the City of Hope, Duarte, California, USA
Protein Identification Using Tandem Mass Spectrometry. Nathan Edwards Informatics Research Applied Biosystems
 Protein Identification Using Tandem Mass Spectrometry Nathan Edwards Informatics Research Applied Biosystems Outline Proteomics context Tandem mass spectrometry Peptide fragmentation Peptide identification
Protein Identification Using Tandem Mass Spectrometry Nathan Edwards Informatics Research Applied Biosystems Outline Proteomics context Tandem mass spectrometry Peptide fragmentation Peptide identification
Answers Chem 250 Unit 1 Proteomics by Mass Spectrometry
 Answers Chem 250 Unit 1 Proteomics by Mass Spectrometry Article #1 Quantitative MS for proteomics: teaching a new dog old tricks. MacCoss MJ, Matthews DE., Anal Chem. 2005 Aug 1;77(15):294A-302A. 1. Questions
Answers Chem 250 Unit 1 Proteomics by Mass Spectrometry Article #1 Quantitative MS for proteomics: teaching a new dog old tricks. MacCoss MJ, Matthews DE., Anal Chem. 2005 Aug 1;77(15):294A-302A. 1. Questions
Purdue-UAB Botanicals Center for Age- Related Disease
 Purdue-UAB Botanicals Center for Age- Related Disease MALDI-TOF Mass Spectrometry Fingerprinting Technique Landon Wilson MALDI-TOF mass spectrometry is an advanced technique for rapid protein identification
Purdue-UAB Botanicals Center for Age- Related Disease MALDI-TOF Mass Spectrometry Fingerprinting Technique Landon Wilson MALDI-TOF mass spectrometry is an advanced technique for rapid protein identification
Chemical Labeling Strategy for Generation of Internal Standards for Targeted Quantitative Proteomics
 Chemical Labeling Strategy for Generation of Internal Standards for Targeted Quantitative Proteomics mtraq Reagents Triplex Christie Hunter, Brian Williamson, Marjorie Minkoff AB SCIEX, USA The utility
Chemical Labeling Strategy for Generation of Internal Standards for Targeted Quantitative Proteomics mtraq Reagents Triplex Christie Hunter, Brian Williamson, Marjorie Minkoff AB SCIEX, USA The utility
Effective Strategies for Improving Peptide Identification with Tandem Mass Spectrometry
 Effective Strategies for Improving Peptide Identification with Tandem Mass Spectrometry by Xi Han A thesis presented to the University of Waterloo in fulfillment of the thesis requirement for the degree
Effective Strategies for Improving Peptide Identification with Tandem Mass Spectrometry by Xi Han A thesis presented to the University of Waterloo in fulfillment of the thesis requirement for the degree
Table of contents Appendix Figure S1 Appendix Figure S2 Appendix Figure S3 Appendix Table S1 Appendix Table S2
 Table of contents Appendix Figure S1 Appendix Figure S2 Appendix Figure S3 Appendix Table S1 Appendix Table S2 1 Appendix Figure S1 Fig. S1. Proteome analysis of the CPE-derived EVs and the choroid plexus
Table of contents Appendix Figure S1 Appendix Figure S2 Appendix Figure S3 Appendix Table S1 Appendix Table S2 1 Appendix Figure S1 Fig. S1. Proteome analysis of the CPE-derived EVs and the choroid plexus
CHEMICAL COVALENT LABELING AND TANDEM MASS SPECTROMETRY FOR TARGETED PROTEIN CHARACTERIZATION, QUANTIFICATION AND STRUCTURAL ANALYSIS YALI LU
 CHEMICAL COVALENT LABELING AND TANDEM MASS SPECTROMETRY FOR TARGETED PROTEIN CHARACTERIZATION, QUANTIFICATION AND STRUCTURAL ANALYSIS By YALI LU A DISSERTATION Submitted to Michigan State University in
CHEMICAL COVALENT LABELING AND TANDEM MASS SPECTROMETRY FOR TARGETED PROTEIN CHARACTERIZATION, QUANTIFICATION AND STRUCTURAL ANALYSIS By YALI LU A DISSERTATION Submitted to Michigan State University in
Targeted protein quantification
 Targeted Quantitative Proteomics Targeted protein quantification with high-resolution, accurate-mass MS Highly selective Very sensitive Complex samples HR/AM A more complete quantitative proteomics picture
Targeted Quantitative Proteomics Targeted protein quantification with high-resolution, accurate-mass MS Highly selective Very sensitive Complex samples HR/AM A more complete quantitative proteomics picture
A Software Suite for the Generation and Comparison of Peptide Arrays from Sets. of Data Collected by Liquid Chromatography-Mass Spectrometry
 MCP Papers in Press. Published on July 26, 2005 as Manuscript M500141-MCP200 A Software Suite for the Generation and Comparison of Peptide Arrays from Sets of Data Collected by Liquid Chromatography-Mass
MCP Papers in Press. Published on July 26, 2005 as Manuscript M500141-MCP200 A Software Suite for the Generation and Comparison of Peptide Arrays from Sets of Data Collected by Liquid Chromatography-Mass
Correction of Errors in Tandem Mass Spectrum Extraction Enhances Phosphopeptide Identification
 pubs.acs.org/jpr Correction of Errors in Tandem Mass Spectrum Extraction Enhances Phosphopeptide Identification Piliang Hao,*,,, Yan Ren,, James P. Tam, and Siu Kwan Sze*,, School of Biological Sciences
pubs.acs.org/jpr Correction of Errors in Tandem Mass Spectrum Extraction Enhances Phosphopeptide Identification Piliang Hao,*,,, Yan Ren,, James P. Tam, and Siu Kwan Sze*,, School of Biological Sciences
Proteomics: the first decade and beyond. (2003) Patterson and Aebersold Nat Genet 33 Suppl: from
 Advances in mass spectrometry and the generation of large quantities of nucleotide sequence information, combined with computational algorithms that could correlate the two, led to the emergence of proteomics
Advances in mass spectrometry and the generation of large quantities of nucleotide sequence information, combined with computational algorithms that could correlate the two, led to the emergence of proteomics
A TMT-labeled Spectral Library for Peptide Sequencing
 A TMT-labeled Spectral Library for Peptide Sequencing by Jianqiao Shen A thesis presented to the University of Waterloo in fulfillment of the thesis requirement for the degree of Master of Mathematics
A TMT-labeled Spectral Library for Peptide Sequencing by Jianqiao Shen A thesis presented to the University of Waterloo in fulfillment of the thesis requirement for the degree of Master of Mathematics
TOMAHAQ Method Construction
 TOMAHAQ Method Construction Triggered by offset mass accurate-mass high-resolution accurate quantitation (TOMAHAQ) can be performed in the standard method editor of the instrument, without modifications
TOMAHAQ Method Construction Triggered by offset mass accurate-mass high-resolution accurate quantitation (TOMAHAQ) can be performed in the standard method editor of the instrument, without modifications
